- Presse
- Blogs und Meer
- Volkszählung im Meer: Mikroorganismen und Meiofauna – Blogpost 6 vom 17. März 2018
Volkszählung im Meer: Mikroorganismen und Meiofauna – Blogpost 6 vom 17. März 2018
Forschungsschiff Sonne, Chile
17. März 2018
Blog #6 Volkszählung im Meer: Mikroorganismen und Meiofauna
Wie Wissenschaftler die Biodiversität der Meere bestimmen, indem sie Taxonomie und Umwelt-DNA miteinander verknüpfen
In einem Teelöffel Sediment leben Milliarden von Mikroben, jede Spezies ist optimal an ihren Lebensraum angepasst. Auf einem einzigen Sandkorn zählten Wissenschaftler bis zu 50000 Bakterien. Andere Arten bevorzugen das Porenwasser in den Zwischenräumen des Sediments oder die Wassersäule darüber. Mit einer Größe von rund ein bis zwei Mikrometern gehören sie zur unsichtbaren Mehrheit unseres Planeten, denn sie machen etwa ein Drittel oder sogar mehr der gesamten Biomasse aus. Trotz ihrer geringen Größe sind diese winzigen Zellen verantwortlich für den Kreislauf der Elemente auf globaler Ebene, da sie die Überreste organischer Materie mineralisieren und Kohlendioxid und Nährstoffe in den Kreislauf zurückführen.
Wenn man auf der Größenleiter von den Bakterien einen Schritt nach oben geht, kommt man zur Meiofauna. Sie deckt mit ihren seltsam aussehenden Organismen den Bereich zwischen 45 Mikrometern und einem Millimeter ab: z.B. Ciliaten, Nematoden und Foraminiferen. Dank ihres Wirkens ist unser blauer Planet voller Leben. Eine der grundlegenden Fragen für Meereswissenschaftler ist, welche Arten dabei wie und wo beteiligt sind.

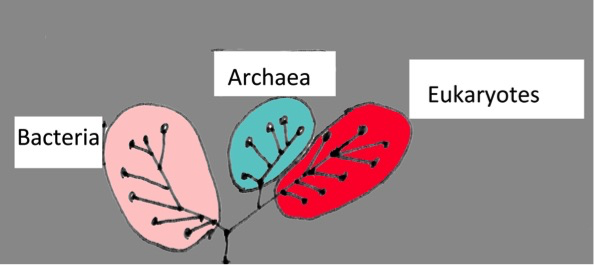
Der Baum des Lebens
Der Baum des Lebens spiegelt die Beziehung zwischen verschiedenen Arten wider. Er kann mit seinen Verästelungen wie eine Ahnentafel betrachtet werden. Wird eine neue Art entdeckt, versuchen Taxonomen, deren Position auf diesem Baum zu bestimmen und ihr einen wissenschaftlichen Namen zu geben. Dabei verwenden sie mehrere Kriterien: z.B. das Aussehen (Morphologie), aber auch Marker wie bestimmte Zellmoleküle. Dies ist eine etablierte Methode und funktioniert gut mit größeren Tieren und Pflanzen. Für kleine Organismen wird es schwieriger, da Mikroorganismen nur in wenigen unterschiedlichen Formen auftreten: z.B. kugel-, stäbchen- und spiralförmig. Zusätzlich wird ihre Bestimmung dadurch erschwert, dass nur 1% der Mikroben in einer Laborkultur gezüchtet werden können, um sie anschließend für biochemische Analysen zugänglich zu machen. Daher haben Wissenschaftler kulturunabhängige Methoden entwickelt, die auf DNA-Analysen beruhen.
Eine taxonomische Datenbank für alle Arten: Die Jagd auf Umwelt-DNA
Viele Wissenschaftler glauben, dass es noch Millionen unentdeckter Arten gibt. Es würde Jahrhunderte dauern, diese mit klassischer Taxonomie zu erfassen, denn um eine mikrobielle Spezies von einer anderen zu unterscheiden, reicht ein Lichtmikroskop nicht aus. Neue Methoden aus der Molekularbiologie bieten einen Ausweg aus dieser Zwickmühle, denn die Arten lassen sich anhand ihrer DNA-Sequenzen unterscheiden. Wie beim genetischen Fingerabdruck der Spuren an einem Tatort, von denen sich die Polizei Hinweise auf den Täter erhofft, setzen Meereswissenschaftler Techniken wie DNA-Sequenzierung ein, um nach ihren Verdächtigen zu suchen. Dabei sind sie sehr ambitioniert und auch erfolgreich. Das große wissenschaftliche Ziel ist es, jede Lebensform auf der Erde zu kategorisieren, um die Taxonomie und den Baum des Lebens zu vervollständigen.
Umwelt-DNA
Neue Techniken, die auf der Analyse von Umwelt-DNA (environmental oder eDNA) beruhen, helfen, den großen Überblick zu bekommen. Da die Natur im Laufe der Evolution gerne ihre eigenen Erfolgsgeschichten kopiert, sind in jeder Art einige wichtige Gene erhalten. Gene sind Bauanleitungen für zelleigene Werkzeuge, die jede Zelle braucht. Natürliche punktuelle Genveränderungen (Mutationen), die die Funktion des Genes nicht beeinträchtigen, haben sich im Laufe der Evolution angesammelt. Diese kleinen Veränderungen der Gensequenzen sind typisch für eine Art. So vergleicht man beim „DNA-Barcoding“ die DNA-Sequenzen eines bestimmten Gens, das in allen untersuchten Arten vorhanden ist. Computeralgorithmen, die auf der Evolution von Arten basieren, berechnen dann die verwandtschaftlichen Beziehungen und konstruieren einen taxonomischen Baum.
Metagenomik
Mit der Metagenomik versuchen Wissenschaftler, einzelne Genome zu rekonstruieren. Das funktioniert auch, wenn im Untersuchungsmaterial verschiedene Mikroorganismen vorhanden sind, wie es bei Umweltproben der Fall ist. Der erste Schritt besteht darin, die Gesamt-DNA zu isolieren. Im nächsten Schritt wird die DNA mittels der Polymerase-Kettenreaktion (PCR) vervielfältigt. Dabei entstehen verschieden lange Kopien bestimmter DNA-Abschnitte, die sie anschließend sequenzieren. Mithilfe von Computerprogrammen suchen sie nach überlappenden DNA-Sequenzen und sortieren und ordnen diese Daten. Im Prinzip beruht diese Methode auf linguistischen Methoden. Es ist, als würde man einen Wust von Textfragmenten verschiedener Schriftsteller miteinander vergleichen und versuchen, die verschiedenen ursprünglichen Texte zu rekonstruieren. Ähnlich wie bei den Autoren hat jede Spezies eine unterschiedliche Präferenz für das „Vokabular“, mit dem sie ihre DNA-Sequenzen kodieren.
Mit dieser Methode können Molekularbiologen auf der Jagd nach Spezies immer mehr „Beute“ machen.
Aber so gut diese Methode zu sein scheint, sie muss stetig optimiert werden. Hier ist das Fachwissen von Taxonomen von entscheidender Bedeutung. Sophie Arnaud-Haond sagt: "Beim Ifremer sind wir an mehreren wissenschaftlichen Projekten über eDNA beteiligt. Wir möchten die Referenzdatenbanken verbessern und all unsere neuen genomischen, taxonomischen, geografischen und Umweltdaten miteinander verknüpfen. Daher ist diese Expedition in den Atacama-Graben sehr wichtig, da hier die Arten sich sehr von denen anderer Standorte unterscheiden können. Wir wollen herausfinden, welche Arten sich auf 8000 Meter Wassertiefe angepasst haben und wer ihre Verwandten sind, die auf der Abyssal-Ebene leben." Daniela Zeppilli stimmt dem zu: "Wir beobachten ähnliche Befunde für einige Meiofauna-Arten. Uns interessiert besonders, ob Nematoden in der Tiefsee genetisch von ihren Vorfahren getrennt sind.“
Mit herzlichen Grüßen von Bord der Sonne
Manfred Schlösser
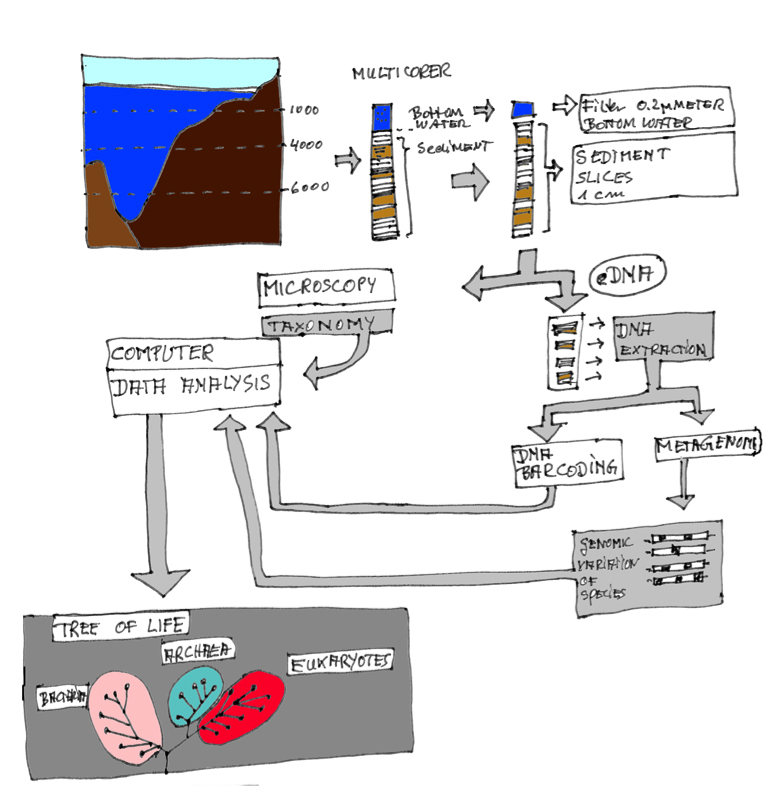
Die Pipeline zur Verknüpfung von Taxonomie- und DNA-Daten. Taxonomie und Genetik sind komplementäre Ansätze, die beide notwendig sind, um die Biodiversität des Meeresbodens zu beschreiben. Der Multicorer sticht mit zylinderförmigen Plexiglasröhren Sedimentproben aus dem Atacama-Graben in 8000 Metern Tiefe. An Bord bearbeiten die Wissenschaftler diese Proben unter sterilen Bedingungen. Zuerst schneiden sie scheibchenweise die 40 Zentimeter langen Kerne in 1 Zentimeter dicke Segmente. Diese werden im Kühlraum fixiert und gelagert. Ein sogenannter Schwesterkern wird separat gelagert und später einer direkten Taxonomie-Prüfung unterzogen. Am Ende der Pipeline haben die neuen Arten ihren Platz im Baum des Lebens.
Meiofauna (Organismen kleiner als ein Millimeter) gefunden in 8000 Metern Tiefe. Von links nach rechts: Ein Kopf eines Polychaetenwurms, ein Nematode, zwei Bärtierchen (a.k.a. Wasserbär), ein Kinorhyncha (Schlammdrache), drei Nematoden. (Skizzen von Manfred Schlösser nach Fotos von Daniela Zeppilli).
Spezifische Fragestellungen dieser Forschungsreise sind:
- Welche sedimentären Prozesse liefern die Nahrung für die hadale Lebensgemeinschaft im Atacamagraben?
- Wie unterscheiden sich der Artenreichtum, die Diversität und die Gemeinschaftsstruktur von Mikroorganismen, der Meio- und Makrofauna im Atacamagraben von Gräben in weniger produktiven Regionen und nahegelegenen Tiefsee- und Schelfgebieten?
- Was sind die generellen biogeochemischen Charakteristika des Oberflächen- und Tiefensediments sowie der Wassersäule im eutrophen Atacamagraben?
- Wie genau verläuft die Mineralisierung bei der Zersetzung organischen Materials im eutrophen Atacamagraben?
- Wie effizient arbeiten die mikrobiellen Gemeinschaften unter extremen hydrostatischen Druckverhältnissen bei der Mineralisierung organischen Materials im Vergleich zu Gemeinschaften in seichteren Gefilden? Und in welchem Ausmaß beeinflussen spezialisierte, bisher unbekannte extremophile mikrobielle Gemeinschaften diese Prozesse?
Weitere Informationen
Mehr Details über das Projekt von der Süddänischen Universität.
Weiteres Bildmaterial zum Projekt.
Bericht über Ronnie N Glud bei Danmarks Radio (auf Dänisch)
Die SONNE ist ein modernes deutsches Forschungsschiff, dessen Fahrtgebiet hauptsächlich im Pazifik liegt. Die Ausfahrt SO261 läuft vom 2. März bis zum 2. April 2018.
Weitere Informationen zur SONNE finden Sie hier.