- Presse
- Pressemeldungen 2020
- Analyse unbekannter Mikroben wird einfacher
Analyse unbekannter Mikroben wird einfacher
Den Wissenschaftlerinnen und Wissenschaftlern aus Bremen ist es mit einer Kombination aus mehreren Methoden gelungen, Eigenschaften von weniger häufigen Organismen einer mikrobiellen Gemeinschaft im Meer über das Metagenom sichtbar zu machen. Das Metagenom sind alle Gene einer mikrobiellen Lebensgemeinschaft zusammengenommen. In dieser Menge an Gen-Informationen wurde die DNA der raren Winzlinge erfolgreich erkannt und im Anschluss analysiert. Das ist relevant, denn: In vielen Lebensgemeinschaften sind ein oder zwei Spezies dominierend und kommen sehr oft vor. Daneben gehören zahlreiche weitere Organismen zur Gemeinschaft, die für die Lebensgemeinschaft zwar auch bedeutsam, aber weniger zahlreich sind. Ihre Rolle zu identifizieren war bisher schwierig.
Im konkreten Fall ging es um Bakterien, die nach Algenblüten vor Helgoland in der Nordsee zu finden sind. „Wir stießen in unseren Proben immer wieder auf eine unbekannte Spezies und wollten mehr über ihre Eigenschaften und ihre Rolle in der Umwelt lernen“, sagt Anissa Grieb vom MPIMM, Erstautorin der Studie. Doch da sich diese Bakterien weder im Labor kultivieren ließen, noch mit herkömmlichen Methoden über das Metagenom zu finden waren, suchte das Team mit Anissa Grieb nach anderen Lösungen.
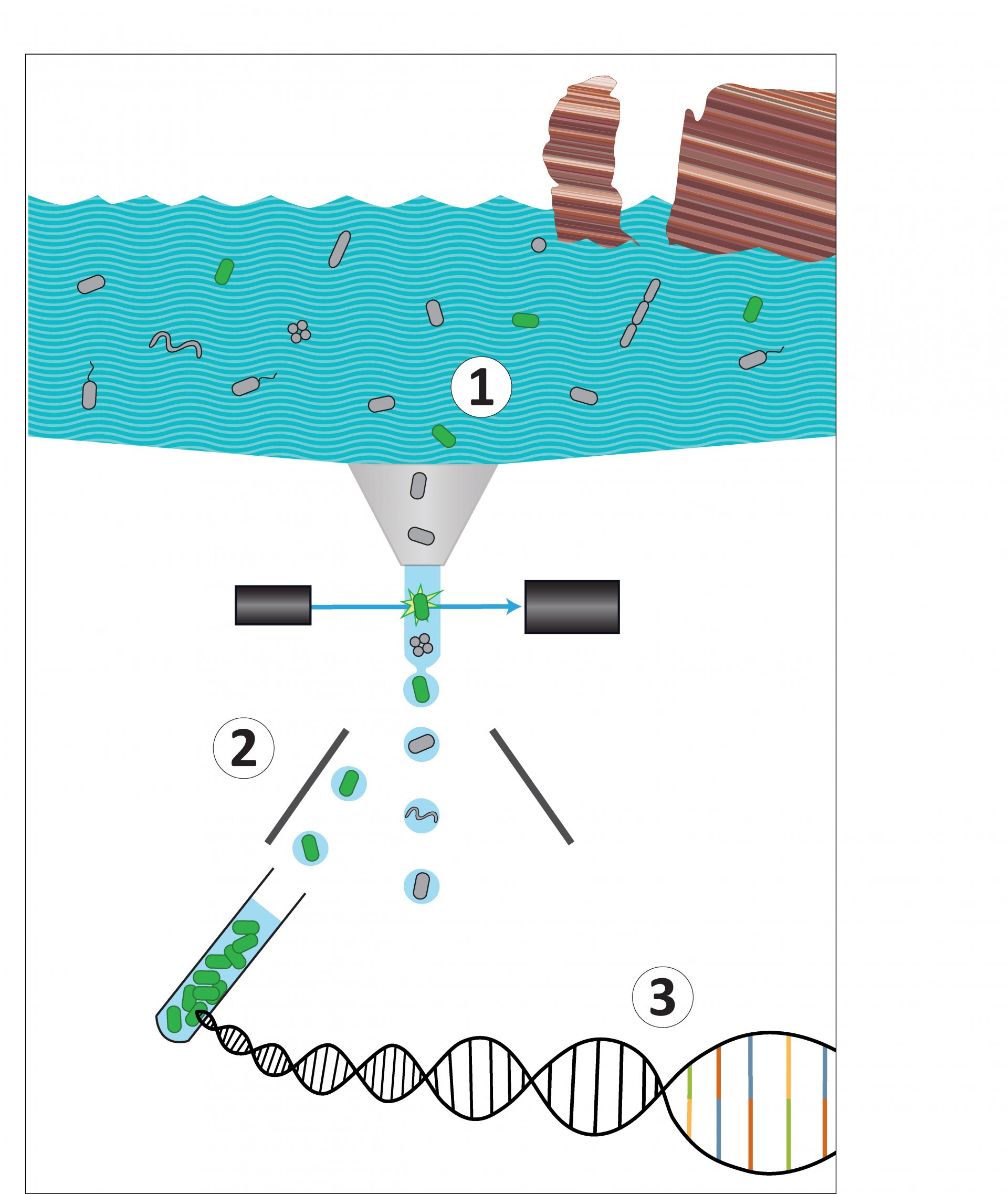
Die Guten ins Töpfchen
Die Idee: „Da wir in unserer Forschungsgruppe über ein Durchflusszytometer mit Zellsortierer verfügen, kam uns der Gedanke, die Zellen der uns interessierenden Spezies vor dem Sequenzieren der DNA auszusortieren“, sagt Grieb. So wird die Diversität geringer und die eigentlich nicht so häufige Spezies kann sich nicht mehr so gut verstecken. Die Wissenschaftlerinnen und Wissenschaftler markierten also mit der FISH-Methode DNA-Abschnitte der Mikroben mit fluoreszierenden Farbstoffen. So können sie die Bakterien erkennen und aussortieren. „Allerdings gab es dabei zwei Herausforderungen: Erstens muss das Farbsignal sehr stark sein, damit wir die Zellen während des Sortierens gut finden können“, sagt Grieb. „Zweitens darf aber die DNA durch den Farbstoff nicht beeinträchtigt werden, da sie für eine qualitativ hochwertige Genom-Analyse im Anschluss möglichst intakt sein muss.“
Gemeinsam mit Forschenden des Joint Genome Institute testete das Team um Anissa Grieb, wie dieser Balanceakt gelingen kann. Erfolg hatten sie am Ende mit einer kürzlich entwickelten Variante der FISH-Methode, und zwar mit der Hybridisierungs-Kettenreaktion (HCR-FISH), die sie durch Anpassung verschiedener Parameter wie der Temperatur für ihre Zwecke optimierten. Zunächst haben sie diese Vorgehensweise mit Reinkulturen im Labor optimiert und anschließend erfolgreich mit den Umweltproben getestet, die vor Helgoland genommen wurden.
Mikroben werden sichtbar
„Wir haben im Ergebnis nun die Möglichkeit, das Genom weniger häufiger Mikroorganismen zu isolieren und dann mit den Standard-Methoden der Gen-Analyse zu untersuchen“, sagt Bernhard Fuchs, Leiter der Forschungsgruppe Durchflusszytometrie am Max-Planck-Institut für Marine Mikrobiologie. „Dadurch können wir die Stoffwechseleigenschaften von Bakterien analysieren, die bislang kaum sichtbar waren.“ So gelang es den Forschenden, die Identität einer weniger häufigen Gattung, deren DNA immer wieder in den Wasserproben aus der Nordsee vor Helgoland auftauchten, zu klären. Oder anders gesagt: Das Versteckspiel ist vorbei.
Originalveröffentlichung
Anissa Grieb, Robert M. Bowers, Monike Oggerin, Danielle Goudeau, Janey Lee, Rex R. Malmstrom, Tanja Woyke and Bernhard M. Fuchs: A pipeline for targeted metagenomics of environmental bacteria. Microbiome 8, February 2020.
DOI: 10.1186/s40168-020-0790-7
Behind the paper: Finding the needle in the haystack via targeted genomics.
Beteiligte Institutionen
- Max-Planck-Institut für Marine Mikrobiologie, Bremen, Deutschland
- DOE Joint Genome Institute, Lawrence Berkeley National Laboratory, Berkeley, USA
Rückfragen bitte an:
Gruppenleiter
Forschungsgruppe Durchflusszytometrie
MPI für Marine Mikrobiologie
Celsiusstr. 1
D-28359 Bremen
|
Raum: |
2222 |
|
Telefon: |


