- Presse
- Pressemeldungen
- Struktur eines rätselhaften Enzyms zur Methanumwandlung
Struktur eines rätselhaften Enzyms zur Methanumwandlung
MCR: Ein Enzym im Herzen des globalen Methankreislaufs
Methan ist ein sehr starkes Treibhausgas – über 25-mal stärker als Kohlendioxid. Deswegen zählt jedes Methanmolekül, das nicht in die Atmosphäre gelangt. Das Enzym MCR hat großen Einfluss darauf, ob Methan in die Luft entkommt oder zu Kohlendioxid umgewandelt wird. Denn MCR ermöglicht sowohl den letzten Schritt der Methanproduktion in Mikroorganismen als den ersten Schritt der anaeroben Methanoxidation — also des Methanverbrauchs in anderen Mikroorgnismen, den ANME.
MCR in methanproduzierenden Archaeen, sogenannten Methanogenen, ist seit Jahrzehnten Gegenstand der Forschung. Darüber, wie es in ANME funktioniert, ist weit weniger bekannt. ANME-Mikroorganismen sind schwer zu erforschen, da sie nicht in Reinkultur im Labor wachsen. Bislang ist nur eine einzige ihrer MCR-Strukturen bekannt, die von einem ANME aus einer mikrobiellen Matte im Schwarzen Meer stammt.
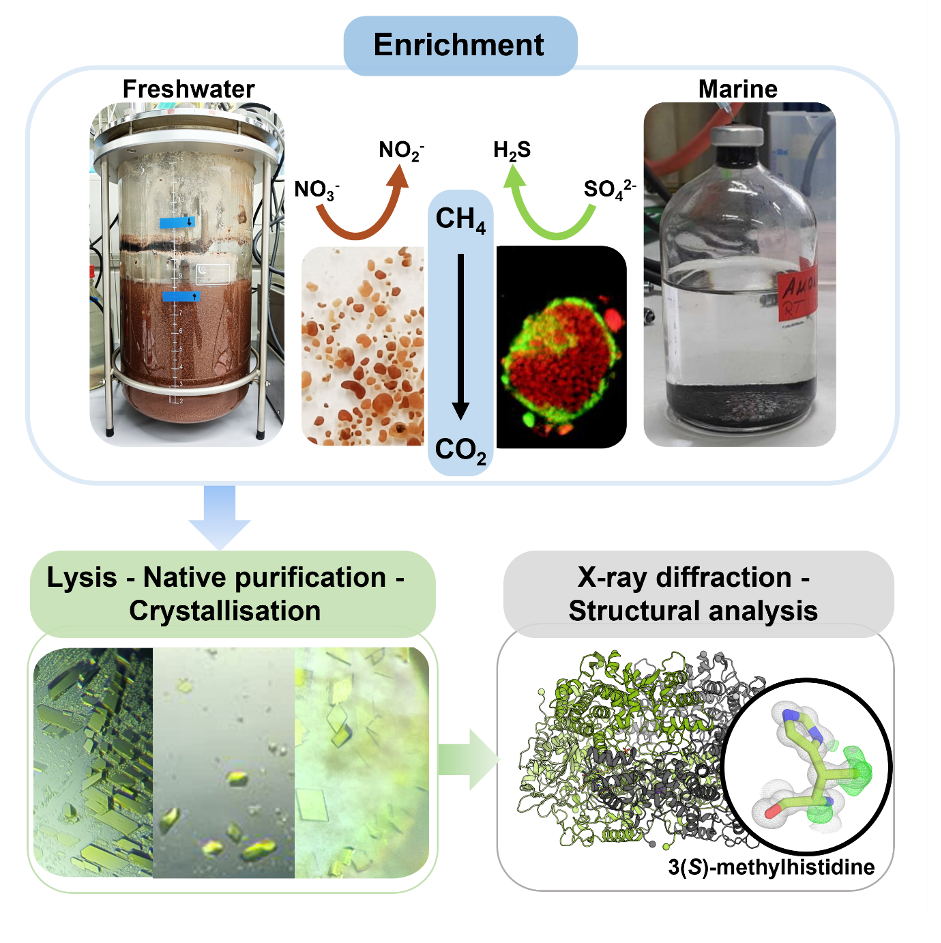
Vom Schlammvulkan zum Bioreaktor: Anreicherung des Unkultivierbaren
“Da sowohl methan-verbrauchende als auch methan-erzeugende Archaeen, also ANME und Methanogene, MCRs brauchen, haben wir uns gefragt, ob diese Enzyme in beide Richtungen gleich funktionieren,“ sagt Projektleiter Tristan Wagner vom Max-Planck-Institut für Marine Mikrobiologie in Bremen (Deutschland). “Um die Unterschiede zwischen den MCRs von ANME und Methanogenen zu verstehen, brauchten wir ein Bild des Enzyms.” Deshalb taten sich die Bremer Forschenden mit Partnerinnen und Partnern aus ganz Europa zusammen, um das Enzym von ANME strukturell zu charakterisieren. Von der Radboud-Universität (Niederlande) kamen mikrobielle Anreicherungen aus zwei verschiedenen Süßwasserproben mit ANME-2d-Archaeen, die Methan mit Hilfe von Nitrat oxidieren, und Gunter Wegener vom Max-Planck-Institut in Bremen lieferte Proben von ANME-2c, die aus Tiefseesedimenten des Amon-Schlammvulkans im Mittelmeer stammen und Konsortien mit sulfatreduzierenden Bakterien bilden.
In diesen Anreicherungen analysierten Forschende der Radboud-Universität und des Nationalmuseums für Naturwissenschaften in Madrid (Spanien) die mikrobiellen Gemeinschaften und lieferten den genomischen Nachweis, dass MCRs vorhanden waren. Wagner und sein Team verarbeiteten die Biomasse aus den Anreicherungen, um die MCRs zu reinigen und Kristalle daraus zu erzeugen.
Ähnliche Enzyme, ungewöhnliche Anpassungen
Unerwarteterweise waren die gelblichen Kristalle, die die Bremer Forschenden aus den Enzymen herstellten, von hervorragender Qualität und lieferten eine Röntgenbeugung auf einem Niveau, das bisher noch nie erreicht wurde. Die folgende Analyse zeigte, dass die MCRs sowohl der Süßwasser- als auch der marinen ANMEs jenen aus schon bekannten methanproduzierenden Archaeen ähnelten – ungeachtet der sehr unterschiedlichen Stoffwechsel der verschiedenen Organismen. „Das stützt unsere Idee, dass Methanproduktion und -oxidation, zwei chemisch gegensätzliche Reaktionen, vom gleichen Enzym ermöglicht werden, das lediglich umgekehrt arbeitet,“ so Wagner. Weitere biophysikalische Experimente an den Kristallen, die an der European Synchrotron Radiation Facility und am Institut de Biology Structurale in Grenoble (France) durchgeführt wurden, bestätigten, dass das MCR aus ANME wohl die gleichen Reaktionsabläufe wie das methanogene Enzym aufweist.
Die Forschenden entdeckten bei dem Enzym eine Rekordzahl von sieben chemischen Veränderungen pro aktiver Stelle. Eine dieser Veränderungen – eine erstmals gesehene Abwandlung namens 3(S)-Methylhistidin, die von einem Experten des Max-Planck-Instituts für terrestrische Mikrobiologie in Marburg (Deutschland) bestätigt wurde – fand sich in der Nähe des wichtigen Nickel-Cofaktors des Enzyms. Diese seltene Veränderung könnte den Archaeen helfen, Methan effizienter abzubauen, ihre genaue Rolle ist aber noch unklar.
ANMEs als Vorbilder für die Klimaforschung
Diese Untersuchung zeigt deutlich, wie wichtig es ist, Enzyme in ihrem natürlichen Umfeld zu verstehen. „Mit nun drei verfügbaren MCR-Strukturen aus ANMEs, darunter das einzige bekannte Enzym mit einem 3(S)-Methylhistidin, können wir besser erforschen, wie die Natur den Methanfluss auf molekularer Ebene in sauerstofffreien Lebensräumen steuert,“ sagt Wagner.
„Indem wir die genutzten Cofaktoren und vorhandenen Veränderungen identifiziert haben, bietet unsere Forschung wertvolle Anregungen für die Entwicklung technischer Systeme, die die natürliche mikrobielle Methanumwandlung nachahmen“, so Wagner abschließend. „Solche Forschungsarbeiten könnten eines Tages dazu beitragen, die Methanfreisetzung zu verringern und die weltweiten Bemühungen um eine emissionsfreie Zukunft unterstützen.“
Originalveröffentlichung
Marie-C. Müller, Martijn Wissink, Priyadarshini Mukherjee, Nicole Von Possel, Rafael Laso-Pérez, Sylvain Engilberge, Philippe Carpentier, Jörg Kahnt, Gunter Wegener, Cornelia U. Welte, Tristan Wagner (2025): Atomic resolution structures of the methane-activating enzyme in anaerobic methanotrophy reveal extensive post-translational modifications. Nature Communications, DOI: 10.1038/s41467-025-63387-1
Beteiligte Institutionen
- Max-Planck-Institut für Marine Mikrobiologie, Celsiusstraße 1, 28359 Bremen, Deutschland
- Abteilung für Mikrobiologie, Radboud Institute for Biological and Environmental Sciences (RIBES), Radboud University, Heyendaalseweg 135, 6525 AJ Nijmegen, Niederlande
- Abteilung für Biogeochemie und mikrobielle Ökologie, Museo Nacional de Ciencias Naturales (MNCN-CSIC), C. de José Gutiérrez Abascal, 2, 28006, Madrid, Spanien
- Univ. Grenoble Alpes, CEA, CNRS, Institut de Biologie Structurale, 71 avenue des Martyrs, 38043 Grenoble, Frankreich
- Europäische Synchrotronstrahlungsanlage, 71 avenue des Martyrs, 38043 Grenoble, Frankreich
- Univ. Grenoble Alpes, CEA, CNRS, IRIG-LCBM UMR 5249, 17 avenue des Martyrs, 38054 Grenoble, Frankreich
- Max-Planck-Institut für Terrestrische Mikrobiologie, Karl-von-Frisch-Strasse 10, 35043 Marburg, Deutschland
- MARUM, Zentrum für Marine Umweltwissenschaften, Universität Bremen, Leobener Straße 8, 28359 Bremen, Deutschland
Rückfragen bitte an:
Gruppenleiter
MPI für Marine Mikrobiologie
Celsiusstr. 1
D-28359 Bremen

Pressereferentin
MPI für Marine Mikrobiologie
Celsiusstr. 1
D-28359 Bremen
|
Raum: |
1345 |
|
Telefon: |

